
Gottfried Wilhelm Leibniz Universität Hannover
Institut für Mensch-Maschine-Kommunikation
FG Graphische Datenverarbeitung
Welfengarten 1 • 30167 Hannover
Ausbildung
Karl-Ingo Friese studierte Mathematik mit Studienrichtung Informatik an der Universität Hannover und schrieb seine Diplomarbeit zum Thema "Randflächenrekonstruktion von Volumenkörpern anhand diskreter Daten". 2002 beendete er sein Studium und arbeitet seitdem als Wissenschaftlicher Mitarbeiter am Lehrstuhl für Graphische Datenverarbeitung, Institut für Mensch - Maschine - Kommunikation der Leibniz Universität Hannover. Dort schrieb er auch seine Promotion mit dem Thema "Entwicklung einer Plattform zur 3D-Visualisierung und -Segmentierung medizinischer Daten" (YaDiV). 2007 war Karl-Ingo Friese als "Organization Chair" verantwortlich für die Planung und Organisation der Cyberworlds 2007 International Conference.
Forschung
Mein Forschungsschwerpunkt liegt im Bereich der Visualisierung, Segmentierung und Analyse medizinischer Daten. Dabei arbeite ich an einer umfangreichen Visualisierungs- und Segmentierungssoftware für DICOM Dateien, die auch Gegenstand meiner Promotion war. Einen besonderer Schwerpunkt bilden dabei Methoden aus der Virtuelle Realität (VR) zur Unterstützung von Arbeitsläufen in der präoperativen Planung.
Aktuelle Projekte
- PhysioLogicHip Konsortium, dass sich mit der Verbesserung der Prozedur von Hüft-OPs beschäftigt. Schwerpunkt des Welfenlab ist dabei die automatische Erkennung der Hüfte unter besonderer Berücksichtigung des Acetabulum und des Femurkopfes sowie die Generierung eines negativen Templates für einen 3D-Drucker.
- Das europäische Marie Curie ITN MultiScaleHuman, dass sich mit der Visualsierung und Analyse von medizinischen Daten aus unterschiedlichen Skalen (von zellular bis Macro-Aufnahmen) und Modalitäten (CT, MRT, Kamera, Messergebnisse) beschäftigt.
- Automatische Erkennung der Orbita (knöcherne Augenhöhle) sowie ein vollautomatischer Formvergleich der gesunden und rekonstruierten Seite für die Kiefer-, Mund- und Gesichtschirurgie.
Einige Bilder aus meiner Forschung:
Lehre
Neben meinen Forschungsinteressen gilt sein besonderes Interesse der Lehre und der Betreuung der Studenten. Regelmäßige stattfindende Lehrveranstaltungen sind z.B. das Labor Graphische 3D Daternverarbeitung in der Medizin, das Programmierpraktikum und die Bildgebenden Systeme für die Medizintechnik.
| Artikel |
| 2016 |
Ricardo Manuel Millán Vaquero, Alexander Vais, Sean Dean Lynch, Jan Rzepecki, Karl-Ingo Friese, Christof Hurschler, Franz-Erich Wolter Helical Axis Data Visualization and Analysis of the Knee Joint Articulation (+video) Journal of Biomechanical Engineering 138. Issue 9. DOI: 10.1115/1.4034005 The American Society of Mechanical Engineers 2016 Beschreibungstext anzeigen We present processing methods and visualization techniques for accurately characterizing and interpreting kinematical data of flexion–extension motion of the knee joint based on helical axes. We make use of the Lie group of rigid body motions and particularly its Lie algebra for a natural representation of motion sequences. This allows to analyze and compute the finite helical axis (FHA) and instantaneous helical axis (IHA) in a unified way without redundant degrees of freedom or singularities. A polynomial fitting based on Legendre polynomials within the Lie algebra is applied to provide a smooth description of a given discrete knee motion sequence which is essential for obtaining stable instantaneous helical axes for further analysis. Moreover, this allows for an efficient overall similarity comparison across several motion sequences in order to differentiate among several cases. Our approach combines a specifically designed patient-specific three-dimensional visualization basing on the processed helical axes information and incorporating computed tomography (CT) scans for an intuitive interpretation of the axes and their geometrical relation with respect to the knee joint anatomy. In addition, in the context of the study of diseases affecting the musculoskeletal articulation, we propose to integrate the above tools into a multiscale framework for exploring related data sets distributed across multiple spatial scales. We demonstrate the utility of our methods, exemplarily processing a collection of motion sequences acquired from experimental data involving several surgery techniques. Our approach enables an accurate analysis, visualization and comparison of knee joint articulation, contributing to the evaluation and diagnosis in medical applications. |
| 2015 |
Ricardo Manuel Millan Vaquero, Sean Dean Lynch, Benjamin Fleischer, Jan Rzepecki, Karl-Ingo Friese, Christof Hurschler, Franz-Erich Wolter Enhanced Visualization of the Knee Joint Functional Articulation Based on Helical Axis Method Bildverarbeitung für die Medizin 2015, Informatik aktuell 2015 449-454 Springer Berlin Heidelberg 2015 Beschreibungstext anzeigen Comprehensive descriptions of the motion in articulating joints open new opportunities in biomedical engineering. The helical axis is a established method that describes flexion-extension at joints, which currently lacks an intuitive visualization. In this paper, we present a comprehensive visualization of knee joint motion based on a direct measurement of the helical axis. The proposed approach incorporates the three-dimensional motion of patient-specific bone segments and the representation of helical axes on the bone, facilitating the observation of the flexion-extension motion at the knee joint. |
Matthias Becker, Karl-Ingo Friese, Franz-Erich Wolter, Nils-Claudius Gellrich, Harald Essig Development of a Reliable Method for Orbit Segmentation & Measuring IEEE International Symposium on Medical Measurements and Applications (MeMeA 2015) 285-290 2015 Beschreibungstext anzeigen Injuries of the bony orbit that, which contains the eye, not only have aesthetic implications but also impair stereoscopic vision. Different techniques are used for surgical treatment. In this work we will present a tool that allows the reliable segmentation of the reconstructed and the unaffected orbit. For this we present a novel anterior closing that follows the clinical understanding of anatomy. To assist in the post-surgical evaluation, we suggest parameters with respect to clinically relevant regions (orbital floor, medial wall) and demonstrate a method to automatically determine them. We evaluate our methods on clinical cases. Date: May, 7, 2015 |
Ricardo Manuel Millan Vaquero, Alexander Vais, Sean Dean Lynch, Jan Rzepecki, Karl-Ingo Friese, Christof Hurschler, Franz-Erich Wolter Helical axis data visualization and analysis of the knee joint articulation (presentation) Accepted as a lecture presentation in 29th International Congress and Exhibition of Computer Assisted Radiology and Surgery (CARS). 2015 Beschreibungstext anzeigen Accepted as a lecture presentation in 29th International Congress and Exhibition of Computer Assisted Radiology and Surgery (CARS 2015). |
Maximilian E. H.Wagner, Nils-Claudius Gellrich, Karl-Ingo Friese, Matthias Becker, Franz-Erich Wolter, Juergen T. Lichtenstein, Marcus Stoetzer, Majeed Rana, Harald Essig Model-based segmentation in orbital volume measurement with cone beam computed tomography and evaluation against current concepts International Journal of Computer Assisted Radiology and Surgery 2015 Beschreibungstext anzeigen Purpose: Objective determination of the orbital volume is important in the diagnostic process and in evaluating the efficacy of medical and/or surgical treatment of orbital diseases. Tools designed to measure orbital volume with computed tomography (CT) often cannot be used with cone beam CT (CBCT) because of inferior tissue representation, although CBCT has the benefit of greater availability and lower patient radiation exposure. Therefore, a model-based segmentation technique is presented as a new method for measuring orbital volume and compared to alternative techniques. Methods: Both eyes from thirty subjects with no known orbital pathology who had undergone CBCT as a part of routine care were evaluated (n = 60 eyes). Orbital volume was measured with manual, atlas-based, and model-based segmentation methods. Volume measurements, volume determination time, and usability were compared between the three methods. Differences in means were tested for statistical significance using two-tailed Student’s t tests. Results: Neither atlas-based (26.63±3.15mm3) nor model-based (26.87±2.99mm3) measurements were significantly different from manual volume measurements (26.65 ± 4.0mm3). However, the time required to determine orbital volume was significantly longer for manual measurements (10.24 ± 1.21 min) than for atlas-based (6.96 ± 2.62 min, p < 0.001) or model-based (5.73 ± 1.12 min, p < 0.001) measurements. Conclusion: All three orbital volume measurement methods examined can accurately measure orbital volume, although atlas-based and model-based methods seem to be more user-friendly and less time-consuming. The new model-based technique achieves fully automated segmentation results, whereas all atlas-based segmentations at least required manipulations to the anterior closing. Additionally, model-based segmentation can provide reliable orbital volume measurements when CT image quality is poor. |
Ricardo Manuel Millán Vaquero, Asan Agibetov, Jan Rzepecki, Marta Ondrésik, Alexander Vais, Joaquim Miguel Oliveira, Giuseppe Patanè, Karl-Ingo Friese, Rui Luís Reis, Michela Spagnuolo, Franz-Erich Wolter A semantically adaptable integrated visualization and natural exploration of multi-scale biomedical data 12th International Conference BioMedical Visualization (MediVis) 2015 19th International Conference on Information Visualisation. IEEE 2015 Beschreibungstext anzeigen The exploration of biomedical data which involves heterogeneous sources coming from different spatial scales and medical domains is a challenging topic in current research. In this work, we combine efforts regarding multi-scale visualiza-tion, multimodal interaction and knowledge formalization for the exploration of multi-scale biomedical data. The knowledge formalization stores and organizes the information sources, the integrated visualization captures all relevant information for the domain expertise of the user and the multimodal interaction provides a natural exploration. We present a concrete example of use of the proposed exploratory system designed for a biologist investigating multi-scale pathologies. |
| 2014 |
Asan Agibetov, Ricardo Manuel Millán Vaquero, Karl-Ingo Friese, Giuseppe Patanè, Michela Spagnuolo and Franz-Erich Wolter Integrated Visualization and Analysis of a Multi-scale Biomedical Knowledge Space Proceedings of the EuroVis Workshop on Visual Analytics 25-29 The Eurographics Association 2014 Beschreibungstext anzeigen The study and analysis of relationships in a complex and multi-scale data set is a challenge of information and scientific visualization. This work proposes an integrated visualization to capture all the important aspects of multi-scale data into the same view by leveraging the multi-scale biomedical knowledge encoded into an underly- ing ontology. Ontology supports visualization by providing semantic means to identify relevant items that must be presented to the user. The study and analysis of relationships across the scales are presented as results of queries to the multi-scale biomedical knowledge space. We demonstrate the prototype of the graphical interface of an integrated visualization framework and the knowledge formalization support in an example scenario related to the musculoskeletal diseases. |
Jan Rzepecki, Ricardo Manuel Millan Vaquero, Alexander Vais, Karl-Ingo Friese, Franz-Erich Wolter Multimodal Approach for Natural Biomedical Multi-scale Exploration Lecture Notes in Computer Science 8888 620–631 Springer Switzerland 2014 Beschreibungstext anzeigen Pathologies which simultaneously involve different spatial scales are often difficult to understand. Biomedical data from different modalities and spatio-temporal scales needs to be combined to obtain an understandable representation for its examination. Despite of requests to improve the exploration of multi-scale biomedical data, no major progress has been made in terms of a common strategy combining the state of art in visualization and interaction. This work presents a multimodal approach for a natural biomedical multi-scale exploration. The synergy of a multi-layered visualization environment based on spatial scales with hand gestures and haptic interfaces opens new perspectives for a natural data manipulation. |
| 2013 |
Roman Vlasov, Karl-Ingo Friese, Franz-Erich Wolter Haptic Rendering of Volume Data with Collision Detection Guarantee Using Path Finding. Transactions on Computational Science 18 Lecture Notes in Computer Science 7848 212-231 2013 Beschreibungstext anzeigen In this paper we present a novel haptic rendering method for exploration of volumetric data. It addresses a recurring flaw in almost all related approaches, where the manipulated object, when moved too quickly, can go through or inside an obstacle. Additionally, either a specific topological structure for the collision objects is needed, or extra speed-up data structures should be prepared. These issues could make it difficult to use a method in practice. Our approach was designed to be free of such drawbacks. An improved version of the method presented here does not have the issues of the original method – oscillations of the interaction point and wrong friction force in some cases. It uses the ray casting technique for collision detection and a path finding approach for rigid collision response. The method operates directly on voxel data and does not use any precalculated structures, but uses an implicit surface representation being generated on the fly. This means that a virtual scene may be both dynamic or static. Additionally, the presented approach has a nearly constant time complexity independent of data resolution. Beschreibungstext verbergen |
Karl-Ingo Friese, Sarah Cichy, Franz-Erich Wolter, Roman Botcharnikov Analysis of tomographic mineralogical data using YaDiV-Overview and practical case study Computers and Geosciences 56 92-103 2013 Beschreibungstext anzeigen We introduce the 3D-segmentation and -visualization software YaDiV to the mineralogical application of rock texture analysis. YaDiV has been originally designed to process medical DICOM datasets. But due to software advancements and additional plugins, this open-source software can now be easily used for the fast quantitative morphological characterization of geological objects from tomographic datasets.In this paper, we give a summary of YaDiV's features and demonstrate the advantages of 3D-stereographic visualization and the accuracy of 3D-segmentation for the analysis of geological samples. For this purpose, we present a virtual and a real use case (here: experimentally crystallized and vesiculated magmatic rocks, corresponding to the composition of the 1991-1995 Unzen eruption, Japan). Especially the spacial representation of structures in YaDiV allows an immediate, intuitive understanding of the 3D-structures, which may not become clear by only looking on 2D-images. We compare our results of object number density calculations with the established classical stereological 3D-correction methods for 2D-images and show that it was possible to achieve a seriously higher quality and accuracy.The methods described in this paper are not dependent on the nature of the object. The fact, that YaDiV is open-source and users with programming skills can create new plugins themselves, may allow this platform to become applicable to a variety of geological scenarios from the analysis of textures in tiny rock samples to the interpretation of global geophysical data, as long as the data are provided in tomographic form. Beschreibungstext verbergen |
| 2012 |
Benjamin Fleischer, Karl-Ingo Friese, Franz-Erich Wolter, Gavin Olender, Christoph Becher and Christoph Hurschler Automatic evaluation of human knee joint kinematics based on MRI images J. Comp. Anim. and Virtual Worlds, Proc. CASA 2012 Wiley 2012 |
Matthias Becker, Karl-Ingo Friese, Franz-Erich Wolter, Nils-Claudius Gellrich and Harald Essig Development of a Reliable Method for Orbit Segmentation & Measuring J. Comp. Anim. and Virtual Worlds, Proc. CASA 2012 Wiley 2012 |
| 2011 |
Karl-Ingo Friese, Philipp Blanke, Franz-Erich Wolter YaDiV - An Open Platform for 3D Visualization and 3D Segmentation of Medical Data The Visual Computer 27 129-139 Springer 2011 Beschreibungstext anzeigen In this work, we present the concept, design and implementation of a new software to visualize and segment 3-dimensional medical data. The main goal was to create a platform that would allow to try out new approaches and ideas while staying independent from hardware and operating system, being especially useful for interdisciplinary research groups. A special focus will be given on fast and interactive volume visualization and a survey on the use of Virtual Reality (VR) and especially haptic/force feedback in medical applications.
Beschreibungstext verbergen |
| Konferenzen |
| 2010 |
YaDiV - The Development of an Open Platform for 3D Visualization and 3D Segmentation of Medical Data 2010 Beschreibungstext anzeigen Keynote Lecture, 3D Anatomical Human Summer School, 2010, Crete Beschreibungstext verbergen |
| Beiträge in Büchern |
| 2014 |
R. M. Millán Vaquero, J. Rzepecki, K.-I. Friese, F.-E. Wolter Visualization and User Interaction Methods for Multiscale Biomedical Data 5 N. Magnenat-Thalmann, O. Ratib, and H. F. Choi Springer London 2014 Beschreibungstext anzeigen The need for handling huge amounts of data from several sources is becoming increasingly important for biomedical scientists. In the past, there were mainly different modalities in imaging techniques that had to be combined. Those modalities usually measured different physical effects from the same object and shared dimensions and resolution. Nowadays, an increasing number of complex use cases exist in biomedical science and clinical diagnostics that require data from various domains, each one related to a different spatiotemporal scale. Multiscale spatial visualization and interaction can help physicians and scientists to explore and understand this data. In the recent years, the number of published articles on efficient scientist-centric visualization and interaction methods has drastically increased. This chapter describes current techniques on multiscale visualization and user interaction and proposes strategies to accommodate current needs in biomedical data analysis. |
| Beiträge in Tagungsbänden |
| 2012 |
Roman Vlasov, Karl-Ingo Friese, Franz-Erich Wolter Ray casting for collision detection in haptic rendering of volume data 2012 Beschreibungstext anzeigen A haptic exploration adds an additional dimension to working with 3D data: a sense of touch. This is especially useful in areas such as medical training and pre-surgical planning, entertainment, CAD and others. Each haptic rendering frame consists of three stages: collision detection, collision response and force- feedback generation. In order to feel the 3D data smoothly, an update rate of at least 1 kHz is required [Brooks Jr. et al. 1990]. Unaddressed practical problems for almost all haptic rendering methods are that no guarantees for collision detection could be given and/or that a special topological structure of objects is required. Here we present an approach which does not have these drawbacks. Furthermore our algorithm has nearly constant time complexity independent of data resolution and does not require any additional pre-computed structures. We focus on volumetric voxel data, since that is the direct input from the scanning devices. Other data types could be transformed to this one, if necessary. Beschreibungstext verbergen |
Roman Vlasov, Karl-Ingo Friese, Franz-Erich Wolter Haptic rendering of volume data with collision determination guarantee using ray casting and implicit surface representation 2012 Beschreibungstext anzeigen Haptic exploration adds an additional dimension to working with 3D data: a sense of touch. This is especially useful in areas such as medical simulation, training and pre-surgical planning, as well as in museum display, sculpting, CAD, military applications, assistive technology for blind and visually impaired, entertainment and others. There exist different surface- and voxel-based haptic rendering methods. Unaddressed practical problems for almost all of them are that no guarantees for collision detection could be given and/or that a special topological structure of objects is required. Here we present a novel and robust approach based on employing the ray casting technique to collision detection, which does not have the aforementioned drawbacks while guaranteeing nearly constant time complexity independent of data resolution. This is especially important for such delicate procedures as pre-operation planning. A collision response in the presented prototype system is rigid and operates on voxel data, and no precalculation is needed. Additionally, our collision response uses an implicit surface representation "on the fly", which can be used with dynamically changing objects. Beschreibungstext verbergen |
| 2008 |
Karl-Ingo Friese, Marc Herrlich, Franz-Erich Wolter Using Game Engines for Visualization in Scientific Applications New Frontiers for Entertainment Computing 2008 Beschreibungstext anzeigen In recent years, the computer gaming industry has become a large and
important market and impressive amounts of money are spent on the development of new game engines. In contrast to their development costs, the price for the final product is very low compared to a professional 3D visualization/animation program. The idea to use this potential for other purposes than gaming seems obvious. This work gives a review on three Serious Gaming projects, analyzes the encountered problems in a greater context and reflects the pros and cons of using game engines for scientific applications in general. Beschreibungstext verbergen |
| 2003 |
Karl-Ingo Friese, Roland Hachmann, Franz-Erich Wolter Content Management Systeme in der Landschaftsplanung 8. internationales Symposion zur Rolle der Informationstechnologien in der und für die Planung sowie zu den Wechselwirkungen zwischen realem und virtuellem Raum, CORP 2003 2003 Beschreibungstext anzeigen Der Einsatz neuer Medien gewinnt auch in der Landschaftsplanung immer mehr an Bedeutung. Dabei ist es wichtig, dass unterschiedliche Projektbeteiligte (z.B. Gemeinden, Verbände, Fachplanungen, etc.) über eine Schnittstelle Zugriff auf gemeinsame Daten erhalten. Im Rahmen eines von der Deutschen Bundesstiftung Umwelt (DBU) geförderten und Mitte 2002 abgeschlossenen Forschungsprojektes wurde am Institut für Landschaftspflege und Naturschutz der Universität Hannover zusammen mit weiteren Projektpartnern eine Informations- und Kommunikationsplattform im Internet aufgebaut. Die positiven aber auch negativen Erfahrungen fliessen in ein weiteres, durch das Bundesamt für Naturschutz (BfN) gefördertes Erprobungs- und Entwicklungsvorhaben, den sogenannten "interaktiven Landschaftsplan" ein.
In beiden Vorhaben handelt es sich um unterschiedlichste Nutzergruppen, die sich in einen Prozess einbringen können und sollen. Als Schnittstelle, die gleichzeitig Fragen des Datenschutzes und der Zugriffsrechte berücksichtigen muss, kommt daher, wie bei fast allen größeren Internetprojekten neueren Datums, ein Content Management System (CMS) zum Einsatz.
Die Erfahrungen aus bereits abgeschlossenen Forschungsprojekten und ein Vergleich freier Public-Domain CMS-Software liessen als Konsequenz nur die Neuentwicklung eines frei verfügbaren Content Management System am Institut für Angewandte Systeme der Universität Hannover zu.
Die Eigenschaften und Möglichkeiten (Nutzer- und Gruppenverwaltung, Bild- und Fotoarchiv, Infobrief, Mailinglisten etc.) des unter dem Arbeitstitel CoMa entwickelten Redaktionssystems und der Dokumentationsbeschreibungssprache CoMaL sollen unter anderem hier aufgezeigt werden. Beschreibungstext verbergen |
| 2000 |
F.-E. Wolter, K.-I. Friese Local & Global Geometric Methods for Analysis Interrogation, Reconstruction, Modification & Design of Shape Proceedings of Computer Graphics International 2000, pp. 137-151, Geneva, Switzerland, IEEE Computer Society, June 2000. Invited paper. (Also available as Welfen Laboratory Report No. 3.) 2000 Beschreibungstext anzeigen This paper gives an overview of some recent methods useful for local and global shape analysis and for the design of solids.
These methods include as new tools for global and local shape analysis the Spectra of the Laplace and the Laplace-Beltrami Operator and the Concept of stable Umbilical Points i.e. stable singularities of the principal curvature line wire frame model of the solid's boundary surface.
Most material in this paper deals with the Medial Axis Transform as a tool for shape interrogation, reconstruction, modification and design. We show that it appears to be possible to construct an intuitive user interface that allows to mould shape employing the Medial Axis Transform.
We also explain that the Medial Axis and Voronoi Diagrams can be defined and computed as well on free form surfaces in a setting where the geodesic distance between two points p, q on a surface S is defined by the shortest surface path on S joining the two points p, q .
This leads to the natural and computable generalized concepts of geodesic Medial Axis and geodesic Voronoi Diagram on free form surfaces. Both can be computed with a reasonable speed and with a high accuracy (of about 12 digits when double floating point arithmetic is used for the computations). Beschreibungstext verbergen |
| Verschiedene |
| 2012 |
Wolter, Rzepecki, Millan, Friese Visualization and Understanding of Medical and Scientific Data 2012 |
| 2015 | ||||||
|
Bachelorarbeit
Hermann Kroll
Leibniz Universität Hannover
Erkennung der Substrukturen des Acetabulum in 3D-Pelvis Modellen
Beschreibungstext anzeigen
Für die Planung von Hüftoperationen ist eine genaue Analyse der patientenindividuellen Geometrie erforderlich. Dabei ist vor allem die Oberflächenstruktur der Hüftpfanne (Acetabulum) als Kontaktregion zum Femurkopf von besonderem Interesse. Betreuer: Karl-Ingo Friese Mai 2015
September 2015
|
||||||
|
Bachelorarbeit
Jakob Riga
Leibniz Universität Hannover
Entwurf und Entwicklung einer haptischen Eingabemethode zur Erfassen von Metainformationen auf 3D-Oberflächen
Beschreibungstext anzeigen
In klinischen Prozessen spielt die tomographische Bildgewinnung heutzutage eine immer größere Rolle. Die dabei gewonnen Schichtbilddaten sind jedoch nur der erste Schritt eines komplexen Verarbeitungsprozesses. Eine auch heute noch schwierige Aufgabe ist die sogenannte Segmentierung, bei der bestimmte Substrukturen (Organe, Knochen, ...) identifiziert werden. Als besonders vielversprechend haben sich dabei Algorithmen gezeigt, die anwendungsspezifisches Modellwissen mit einbeziehen (modelbasierte Segmentierung). Betreuer: Karl-Ingo Friese Juli 2015
September 2015
|
||||||
|
Masterarbeit
Daniel Busse
Leibniz Universität Hannover
Entwicklung eines stochastischen Modells für die 3D-Segmentierung medizinischer Tomographiedaten
Beschreibungstext anzeigen
Die Medizin hat im letzten Jahrhundert in Form von bildgebenden Verfahren ein bedeutendes Hilfsmittel erhalten. Basierend auf verschiedenen physikalischen Eigenschaften, bilden sie die innere Anatomie eines Patienten auf diskrete Volumendaten ab: Es entstehen dreidimensionale Bilder, auf denen unterschiedliche Gewebearten durch variierende Grauwerte dargestellt werden. Betreuer: Karl-Ingo Friese Januar 2015
Juli 2015
|
||||||
| 2014 | ||||||
|
Bachelorarbeit
Jurek Leonhardt
Automatische Erkennung des Acetabulum in 3D-Pelvis Modellen
Beschreibungstext anzeigen
Hüftimplantate zählen zu den häufigsten orthopädischen Operationen. Für eine patientenindividuelle Planung von Implantat und Eingriff ist es notwendig, sowohl die Hüfte (Pelvis) als auch den Oberschenkelknochen (Femur) zu identifizieren. Von besonderem Interesse ist dabei die Kontaktregion, die aus dem Kopf des Femur (caput ossis femoris) und der Hüftpfanne (acetabulum) besteht.
Ziel dieser Arbeit ist es, das Acetabulum in gegebenen 3D-Pelvis Modellen zu identifizieren. Dafür sollen unterschiedliche Algorithmen untersucht, implementiert und verglichen werden. Betreuer: Karl-Ingo Friese Mai 2014
September 2014
|
||||||
| 2013 | ||||||
|
Masterarbeit
Friedrich Hattendorf
Implementation und Analyse von Triangle Strip Algorithmen
Beschreibungstext anzeigen
Volumenkörper werden in der Computergrafik oft durch die Darstellung ihrer Oberfläche als Dreiecksnetz repräsentiert. Gerade bei komplexeren Objekten kann dabei die Dreieckszahl schnell mehrere Millionen erreichen, was auch heute noch eine Herausforderung für die Grafikkarte darstellt. Zwar existieren Verfahren, die ein Dreiecksnetz vereinfachen (= die Anzahl der Dreiekce reduzieren), dabei besteht jedoch immer die Gefahr, dass wichtige Details der Oberfläche Verloren gehen können oder schlimmstenfalls sogar Selbstüberschneidungen erzeugt werden. Eine interessante Alternative stellen die sogenannten Triangle-Strips dar. Bei dieser Datenstruktur, wird versucht, das Netz aus möglichst langen Dreiecks-Streifen zusammenzusetzen, bei der sich jeweilst zwei aufeinanderfolgende Dreiecke genau eine Kante teilen. Durch diesen Trick lässt sich im Idealfall der benöitgte Speicheraufwand auf 1/3 reduzieren. Das Problem der Findung einer solchen idealen Zerlegung ist jedoch N(P) vollständig. Daher existieren eine Vielzahl von Verfahren, die mit heuristischen Methoden versuchen, eine möglichst gute Triangle Strip Darstellung zu finden. Ziel dieser Arbeit ist es, verschiedene Verfahren aus dieser Kateogrie zu implementieren und für Dreiecksflächen aus medizinischen Anwendungen zu vergleichen. Zusätzlich soll ein eigenes Verfahren implentiert werden, bei dem der Augenmerk nicht in einer möglichst guten Strip Darstellung sondern der Erzeugung der Strip Struktur ohne nennenswerten Zeit- und Speicheraufwand liegt. Betreuer: Karl-Ingo Friese November 2012
Mai 2013
|
||||||
| 2012 | ||||||
|
Masterarbeit
Marco Köhnen
Entwicklung einer halbautomatischen Segmentierungsmethode zur Bestimmung der intrakraniellen Luftmenge nach einer Schädeloperation
Beschreibungstext anzeigen
In dieser Arbeit soll ein neuer Ansatz automatischer Verfahren mit Hilfe von Deformable Models und gängiger Segmentierungsverfahren verfolgt werden, um den Liquorverlust nach einer Schädel-Operation zu Bestimmen.
Beschreibungstext verbergen
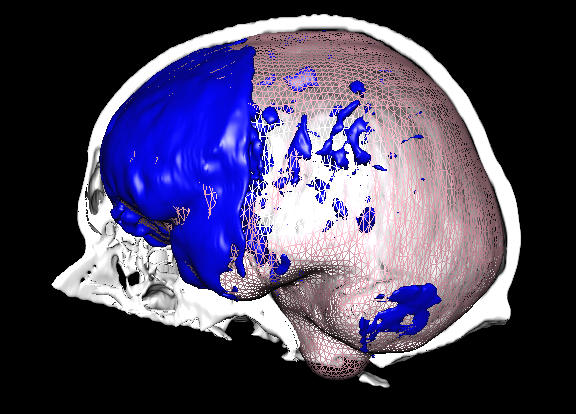 Hintergrund sind Operationen, bei denen der Patient in einer halb-sitzenden Stellung am Hinterkopf operiert wird. Eine Folge davon ist ein Verlust an Flüssigkeit, die sich im Laufe der Zeit rückbildet. Um diesen Rückbildungsprozess untersuchen zu können, soll ein halb-automatisches Verfahren zur Bestimmung der intrakraniellen Luftmenge etnwickelt und mittels umfangreicher bereits vorliegender Daten auf Reproduzierbarkeit und Güte getestet werden.
Hintergrund sind Operationen, bei denen der Patient in einer halb-sitzenden Stellung am Hinterkopf operiert wird. Eine Folge davon ist ein Verlust an Flüssigkeit, die sich im Laufe der Zeit rückbildet. Um diesen Rückbildungsprozess untersuchen zu können, soll ein halb-automatisches Verfahren zur Bestimmung der intrakraniellen Luftmenge etnwickelt und mittels umfangreicher bereits vorliegender Daten auf Reproduzierbarkeit und Güte getestet werden.
Betreuer: Karl-Ingo Friese Juni 2012
Dezember 2012
|
||||||
|
Bachelorarbeit
Peter Denis
Stereographische Visualisierung von 360° Aufnahmen auf einem Head Mounted Display (HMD)
Beschreibungstext anzeigen
Seit einiger Zeit sind Panoramafotos ein beliebtes Mittel zur Illustration von öffentlichen Plätzen oder Produkten geworden. Klassischerweise wird dabei eine normale Kamera rotiert und die dabei entstehenden Einzelaufnahmen zu einem großen Panorama zusammengefügt. Projiziert man dieses Panorambild zusätzlich auf eine zylindrische Geometrie, entsteht der Eindruck, als ob man sich in der Szene befindet.
Beschreibungstext verbergen
Betreuer: Karl-Ingo Friese Mai 2012
September 2012
|
||||||
|
Bachelorarbeit
Marc Lühr
Interaktive Szeneexploration mittels Datenhandschuh und optischem Tracking
Beschreibungstext anzeigen
Im alltäglichen Leben ist die Hand einer der wesentlichsten Werkzeuge um mit der umgebenden Welt zu interagieren. Diese Interaktionsmöglichkeit wird bei der Arbeit mit Computern nur unzureichend über eingaben via Tastatur und Maus umgesetzt. Besonders bei Interaktionen mit einer dreidimensionalen Welt sind diese limitierten Werkzeuge ein großes Hinderniss für eine intuitive Steuerung. Natürlicher wäre eine Steuerung die die Bewegung der Hände mit einbeziehen würde. Dazu soll in dieser Bachelorarbeit der CyberGlove 2 Datenhandschuh zur Exploration einer dreidimensionalen Szene verwendet werden. Die Fingerstellung wird dabei über Widerstandssensoren im Handschuh gemessen und kann so zur Darstellung des Handmodells verwendet werden. Die Informationen über die Lage der Hand im Raum soll anschließend, durch am Kopf befestigte Kameras und Marker auf der Hand, rekonstruiert werden. Diese Arbeit soll in ein plattformunabhängiges Java-Framework integriert werden, dass die Darstellung dreidimensionaler Szenen mittels eines Head-Mounted-Displays ermöglicht soll. Die Darstellung wird in der Bachelorarbiet "Stereographische Visualisierung von 360° Aufnahmen auf einem Head Mounted Display (HMD)" durch Peter Denis implementiert. Durch die stereoskopsiche Darstellung der Szene gepaart mit der Interaktion durch die Hand soll so eine hohe Immersion erreicht werden, die ein intuitives Arbeiten in der virtuellen Szene erlaubt. Betreuer: Karl-Ingo Friese Mai 2012
September 2012
|
||||||
|
Diplomarbeit
Benjamin Fleischer
Welfenlab, IMS
Halbautomatische Segmentierung der knöchernen Strukturen des Kniegelenks in MRT Aufnahmen
Beschreibungstext anzeigen
Zuverlässige Segmentierung ist eine der wichtigsten und zugleich komplexesten Aufgaben in der modernen Volumen Analyse. Besonders im Bereich der Medizin sind bildgebende Verfahren heute ein integraler Bestandteil des klinischen Alltags geworden, was zur Folge hat das ein hoher Prozentsatz der Arbeitszeit auf die Nachbearbeitung bzw. Strukturfindung aufgewendet werden muss. Hier setzen (teil-) automatisierte, modellbasierte Verfahren an. Eine prominente Klasse der modellbasierten Segmentierungsverfahren ist der "Deformable Model" Ansatz. Grundidee ist die Verwendung eines initiales Modells, üblicherweise durch ein 3D Netz repräsentiert, welches in einem iterativen Verfahren an die zu findende Kontur angepasst wird und sich in jedem Schritt ausbreitet oder schrumpft, vergleichbar mit einem Ballon in den (lokal) Luft hingepumpt oder abgelassen wird. Ziel dieser Arbeit ist es, unter Verwendung des am Welfenlab entwickelte YaDiV Deformable Model Framework (YDMF) angepasste Modelle für die knöchernen Strukturen im Bereich des menschlichen Kniegelenks (Patella, Femur, Tibia) zu entwickeln. Als Volumendaten werden MRT-Aufnahmen zum Einsatz kommen, was eine zusätzliche Herausforderung an das Modell darstellt, da die Differenzierung von knöchernen Strukturen durch Weichgewebe in MRT-Aufnahmen erschwert wird.
Als erster Schritt soll ein Modell für die Patella entwickelt werden, welches mit anatomischen Landmarken versehen wird und durch eine lokal und global angepassten Expansions- bzw. Erosionskraft die Strukturfindung ermöglicht. Das Modell soll mit einer geringen Auswahl an Datensätzten trainiert und anschließend durch die Anwendung in neuen Aufnahmen evaluiert werden. Die gewonnen Erkenntnisse sollen dann für die Modellierung der Femur bzw. Tibia Modelle genutzt werden, bei denen die zusätzliche Herausforderung besteht, das hier oft nur eine Teilabdeckung im Aufnahmebereich besteht. Betreuer: Karl-Ingo Friese November 2011
Mai 2012
|
||||||
| 2011 | ||||||
|
Studienarbeit
Friedrich Krüger
Dynamische Erzeungung von Triangle Strips als Erweiterung des Marching Cube Verfahrens
Beschreibungstext anzeigen
Ein gängiges Verfahren zur indirekten Visualisierung von Volumendaten ist der Marching-Cube Algorithmus. Dabei wird jeweils ein (wandernder) Würfel über die Volumendaten gelegt und die Daten an dessen Eckpunkte ausgewertet. Je nach Eckpunktkonfiguration wird eine bereits vorberechnete Dreiecksmenge aus einer Lookup-Tabelle mit 256 Einträgen verwendet.
Ein Manko diese Verfahrens ist es, dass selbst bei kleinen Volumendaten sehr schnell große Dreiecksmengen entstehen können. Eine nachträgliche Reduzierung würde jedoch die Laufzeit beeinträchtigen. Ein Königsweg kann hier die Verwendung von sogenannten Triangle-Strips darstellen. Bei dieser optimierten Repräsentation von Dreiecksflächen lässt sich nicht nur der Memory-Footprint reduzieren, sondern auch die Darstellungsqualität (FPS) erheblich verbessern.
Daher ist es das Ziel dieser Studienarbeit, die Triangle Strips ohne Verlust der Performance dynamisch zu erzeugen.
Beschreibungstext verbergen
Betreuer: Karl-Ingo Friese September 2011
Dezember 2011
|
||||||
|
Bachelorarbeit
Sergi Lazaro
Modelling of realistic Blood Vessel Geometry
Beschreibungstext anzeigen
With modern laser technology it has become possible to print living 3D organic structures like organs or muscles. Currently one of the main problems of this procedure is that the living material dies off during the printing process, therefor it becomes necessary to generate a supply.
The goal of this thesis is to analyse and construct the geometry of blood vessels. A vessel consists of an inner and outer diameter and may branch several times. A special focus will lie on the two end points, which function as input and output of the liquid supply system.
For this it is planned, to construct the basic structure as a connected graph generated by a Lindenmayer System. This graph is then understood as the medial axis of the vessel geometry. The inner and outer vessel diameter define the radius function. For the construction process, it should be possible to tweak these parameters.
Beschreibungstext verbergen
Betreuer: Karl-Ingo Friese Mai 2011
September 2011
|
||||||
|
Bachelorarbeit
Mona Lepke
Untersuchung zur Form- und Mustererkennung auf LC Displays bei benachbarten Farbwerten
Beschreibungstext anzeigen
In vielen Anwendungen wird Information als Farbwert kodiert. So stellt z.B. eine Wärmekamera Temperaturwerte in einer Falschfarbenskala dar. In der Medizin werden Intensitätswerte oft als Graustufen repräsentiert. Dabei können unterschiedliche physikalische Effekte kodiert werden, bei einem CT Bild stehen die Graustufen für die Schwächung der Röntgenenergie, bei einem MRT Bild für die Kernspinrelaxationszeit. Eine hohe Intensität wird dabei meist als Weiss, eine niedrige als Schwarz dargestellt, benachbarte Intensitäten erhalten ähnliche oder (bedingt durch den eingeschränkten Farbraum) sogar die gleiche Grauwerte.
Beschreibungstext verbergen
Oft übersehen wird dabei die Frage, ob die darstellbaren Farb- oder Grauwerte auch optisch auseinandergehalten werden können. Im DICOM Standard wurden dazu umfangreiche Untersuchungen durchgeführt und für die medizinische Befundung spezielle Monitore und Kalibrierungstechniken gefordert. Wie aber sieht es auf normalen LCD Monitoren aus, die z.B. in der präoperativen Planung verwendet werden? Hier setzt diese Arbeit an, in der Benutzertests zur Form- und Mustererkennung durchgeführt werden sollen. Dabei soll auf den Ergebnissen der Studienarbeit von Lara Toma aufgebaut werden, die ebenfalls am Welfenlab entstanden ist.Betreuer: Karl-Ingo Friese Mai 2011
September 2011
|
||||||
|
Masterarbeit
Matthias Becker
Welfenlab + Kiefer-, Mund und Geschitschirurgie der MHH
Modellbasierte Orbita Segmentierung und die automatisierte Bestimmung anatomisch relevanter Parameter
Beschreibungstext anzeigen
Der Begriff Segmentierung beschreibt die Auswahl von Voxeln in einem Volumendatensatz entsprechend einem vom Benutzer gewählten Homogenitätskriterium. Ein einfaches Verfahren könnte z.B. alle Voxel zwischen einer unteren oder oberen Intensitätsschranke segmententieren (Min-Max-Verfahren). Sollen komplexere Formen erkannt werden, kommen meist modellbasierte Verfahren zum Einsatz. Ein Beispiel für eine solche, komplexe Form, stellt die knöcherne Augenhöhle (Orbita) dar. Ziel dieser Masterarbeit war es, modellbasierte Verfahren anhand des klinisch relevanten Beispiels der Orbita-Segmentierung zu untersuchen. Die entwickelten Algorithmen sind jedoch allgemeiner und auch für andere Anwendungsfälle einsetzbar. Ein Hauptaugenmerk lag dabei auf dem Deformable Model Ansatz. Dabei wird ein zur gesuchten Segmentkontur topologisch äquivalentes Startmodell schrittweise "aufgeblasen" und durch geeignete innere oder äußere Parameter (Formerhaltung, Gradienten, ...) gebremst. Zur Demonstration des Verfahrens wurde ein Modul für die von Karl-Ingo Friese geschriebene Software YaDiV entwickelt werden, das die Visualisierung und Kontrolle der Evolutionssparameter in jedem Schritt erlaubt. In der Ausarbeitung wurde der Einfluss unterschiedlicher Parametergewichtungen untersucht. Ein weiteres Ziel dieser Arbeit bestand in der automatisierten Bestimmung anatomisch interessanter Punkte und Größen (Winkel, Abstände, etc.) und einer Analyse der Segmentgüte. Betreuer: Karl-Ingo Friese Oktober 2010
April 2011
|
||||||
|
Studienarbeit
Benjamin Fleischer
IMS + Welfenlab
Entwicklung eines Moduls zur automatisierten Bestimmung der Kniegelenkskinematik und -biomechanik durch dreidimensionale in vivo-Analyse mittels MRT
Beschreibungstext anzeigen
Durch nichtinvasive Verfahren gewonnene 3D-Bilddaten finden auch im Bereich der Kniegelenkserkrankungen und -verletzungen eine immer bereitere Anwendung. Durch Techniken wie dem (offenen) Up-Right MRT ist es möglich, funktionelle Aufnahmen in unterschiedlichen Gelenkstellungen mit und ohne (natürliche) Belastung durchzuführen.
Von besonderem klinischen Interesse ist die Bestimmung biomechanischer Parameter, wie z.B. dem Patello-femoralen Winkel zwischen der Kniescheibe und der Hauptachse des Oberschenkelknochens. Dabei wird das Ziel verfolgt, bestehende oder zukünftige nicht-physiologische Verhältnisse besser
Beschreibungstext verbergen
zu quantifizieren und Behandlungen objektiver bewerten zu können. Für die Bewältigung dieser Aufgabe ist eine intensive interdisziplinäre Zusammenarbeit (Medizin, Mathematik, Informatik, ...) erforderlich. Derzeit ist die Bestimmung dieser Größen nur bedingt automatisiert und erfordert viel Zeit durch medizinisch ausgebildete Anwender. Bereits existierende Ansätze haben weitestgehend prototypischen Charakter. Für den Einsatz in der klinischen Praxis ist es jedoch erfoderlich, ein weitestgehend automatisiertes Verfahren (wenn möglich ohne die Verwendung mehrerer Softwarepakete) zu entwickeln, das die biomechanischen Parameter in vertretbarer Zeit berechnet. Dazu soll das Verfahren als Modul für die von Karl-Ingo Friese am Lehrstuhl für Graphische DV der LUH ("Welfenlab") entwickelte Software YaDiV integriert werden. Die Arbeit wird durch das Labor für Biomechanik und Biomaterialien (LBB) der Orthopädischen Klinik begleitet, das die Betreuung für die medizinischen Fragen übernimmt. Datengrundlage sind dabei die bereits in Patella, Femur und Tibia vorsegmentierten Volumendaten, die durch das LBB zur Verfügung gestellt werden. In dieser Arbeit sollen bereits bestehende Verfahren analysiert, verglichen und durch eigene Ansätze verbessert werden. So soll z.B. die Parameterbestimmung unabhängig von der Lage der drei Strukturen im Aufnahmeraum möglich sein. Betreuer: Karl-Ingo Friese August 2010
Februar 2011
|
||||||
| 2010 | ||||||
|
Studienarbeit
Johannes Wahle
Illustrative Volumendarstellung mit Lit Sphere Mapping unter Berücksichtiung von vorhandenen Segmentinformationen
Beschreibungstext anzeigen
Ray Casting ist eine weit verbreitete Methode, Volumendaten, bswp. aus der Medizin, zu visualisieren. Bildlich beschrieben lautet die Grundidee, einen imaginären Strahl durch jeden Pixel des zu berechnenden Bildes zu verfolgen, der ausgehend vom Betrachter den Volumendatensatz durchläuft und dabei einen Farbeindruck erzeugt. Das Verfahren ist somit - nicht nur namentlich - eng verwandt mit Konzepten aus dem Bereich Ray Tracing.
Neben der möglichst realistischen Darstellung mit etablierten Lichtmodellen wie Goraud- oder Phong-Shading gibt es auch weniger bekannte Modelle, die einen eher künstlerischen Aspekt der Volumendarstellung verfolgen. Eines dieser Modelle ist das sogenannte Lit-Sphere-Mapping. Das Konzept geht zurück auf die Arbeit von Sloan et al. aus dem Jahr 2001, wo die Autoren ein Verfahren vorschlugen, mit dem sich Schattierungstechniken, wie sie in der Kunst verwendet werden, vergleichsweise einfach in Rendering Verfahren integrieren lassen. Künstler verwenden Schattierungs-Skizzen in Form einer projizierten Halbkugel, um in ihrem Bild einen bestimmten Zeichenstil zu verwenden. Dabei wird die Farbschattierung als Textur auf einer Kreisscheibe aufgetragen, die alle möglichen Normalen der Halbkugel als Stil- bzw. Farbinformation enthält.
Ein Problem der Bilderzeugung mit einer intensitätswertbasierten Transferfunktion ist, dass unterschiedliche Gewebe (bedingt durch das verwendete bildgebende Verfahren wie CT, MRT, PET, ...) einen ähnlichen oder identischen Wert besitzen können. Ziel dieser Arbeit ist es, bereits vorhandene Segmentinformationen in der Farbgebung zu berücksichtigen. Dazu sollen unterschiedliche Ansätze implementiert und als Module für die die am Lehrstuhl entwickelte YaDiV-Plattform integriert und verglichen werden.
Beschreibungstext verbergen
Betreuer: Karl-Ingo Friese Mai 2010
August 2010
|
||||||
| 2009 | ||||||
|
Diplomarbeit
Robert Christian Meyer
Welfenlab
Entwurf und Implementierung eines atlasbasierten Segmentierungsverfahrens für medizinische Volumendaten
Beschreibungstext anzeigen
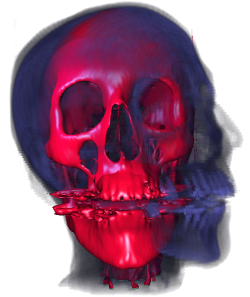 In der medizinischen 3D Datenverarbeitung versteht man unter dem Begriff Segmentierung die Identifikation von medizinisch relevanten Strukturen, wie z.B. Organen, Knochen oder pathologischen Veränderungen.
Bei der modellbasierten Segmentierung wird ein Modell der im Datensatz dargestellten Körperregion zur Verfügung gestellt, das an eine zu segmentierende Aufnahme angepasst werden kann. Die Idee dieses Verfahrens ist für medizinische Anwendungen besonders interessant, weil hier sehr oft sehr ähnliche Datensätze segmentiert werden. Diese Variante der Segmentierungsverfahren wird als Atlasbasierte Segmentierung und die als Modell genutzte Aufnahme als Atlas bezeichnet.
Die größte Schwierigkeit dieses Verfahrens liegt darin, die Segmente des Atlas korrekt auf den Datensatz abzubilden. Da zwischen zwei Aufnahmen immer Differenzen auftreten, die beispielsweise durch die Aufnahme selbst (z.B. leicht unterschiedlicher Blickwinkel) oder auch durch eine leicht veränderte Form der betrachteten Struktur (z.B. beim Vergleich eines gewachsenen Tumors) ergeben können, muss eine Transformation gefunden werden, der den Atlas möglichst genau auf den Datensatz abbildet. Allgemein wird dieser Vorgang Registrierung genannt und ist Gegenstand vieler aktueller Forschungsvorhaben. Dabei wird zwischen einer affinen und einer elastischen Registrierung unterschieden. Im regiden Teil werden die Volumendaten durch eine affine Transformation (Verschiebung, Rotation, Skalierung, ...) vorregistriert. In der elastischen Registrierung werden dann durch eine auf B-Splines basierende multigrid Deformation lokale Differenzen ausgeglichen.
Ziel dieser Arbeit ist es, ein aktuelles atlasbasiertes Segmentierungsverfahren zu implementieren und in die bestehende YaDiV Plattform zu integrieren. In der medizinischen 3D Datenverarbeitung versteht man unter dem Begriff Segmentierung die Identifikation von medizinisch relevanten Strukturen, wie z.B. Organen, Knochen oder pathologischen Veränderungen.
Bei der modellbasierten Segmentierung wird ein Modell der im Datensatz dargestellten Körperregion zur Verfügung gestellt, das an eine zu segmentierende Aufnahme angepasst werden kann. Die Idee dieses Verfahrens ist für medizinische Anwendungen besonders interessant, weil hier sehr oft sehr ähnliche Datensätze segmentiert werden. Diese Variante der Segmentierungsverfahren wird als Atlasbasierte Segmentierung und die als Modell genutzte Aufnahme als Atlas bezeichnet.
Die größte Schwierigkeit dieses Verfahrens liegt darin, die Segmente des Atlas korrekt auf den Datensatz abzubilden. Da zwischen zwei Aufnahmen immer Differenzen auftreten, die beispielsweise durch die Aufnahme selbst (z.B. leicht unterschiedlicher Blickwinkel) oder auch durch eine leicht veränderte Form der betrachteten Struktur (z.B. beim Vergleich eines gewachsenen Tumors) ergeben können, muss eine Transformation gefunden werden, der den Atlas möglichst genau auf den Datensatz abbildet. Allgemein wird dieser Vorgang Registrierung genannt und ist Gegenstand vieler aktueller Forschungsvorhaben. Dabei wird zwischen einer affinen und einer elastischen Registrierung unterschieden. Im regiden Teil werden die Volumendaten durch eine affine Transformation (Verschiebung, Rotation, Skalierung, ...) vorregistriert. In der elastischen Registrierung werden dann durch eine auf B-Splines basierende multigrid Deformation lokale Differenzen ausgeglichen.
Ziel dieser Arbeit ist es, ein aktuelles atlasbasiertes Segmentierungsverfahren zu implementieren und in die bestehende YaDiV Plattform zu integrieren.Betreuer: Karl-Ingo Friese Juni 2009
Dezember 2009
|
||||||
|
Masterarbeit
Marc Christoph Vollmer
Leibniz Universität Hannover
Entwurf und Implementation einer haptischen Schnittstelle für YaDiV
Beschreibungstext anzeigen
In der heutigen Zeit wird aufgrund neuer technischer Innovationen und verstärkter Rechenleistung das Immersionsgefühl des Benutzer immer weiter gesteigert. Das Teilgebiet zur Integration von Haptik in eine dreidimensionale virtuelle Welt ist ein wesentlicher Bestandteil davon. Im Rahmen dieses Forschungsgebiet werden Algorithmen entwickelt mit dessen Hilfe Objekte nicht nur visuell begreifbar, sondern tatsächlich erfühlbar werden.
Im Rahmen dieser Arbeit werden verschiedene Ansätze zur Anwendung von Haptik auf virtuelle Objekte untersucht. Dazu gehören Verfahren zur Kollisionerkennung sowie die Berechnungsmethoden für rückwirkende Kräfte. Zusätzlich werden noch Algorithmen zur Deformation in Echtzeit untersucht. Darunter fallen elastische Deformationen beim Kontakt mit dem virtuellen Objekt und Algorithmen zur permanenten Verformung der Objekte.
Alle Ergebnisse wurden abschließend in ein Programm für die Visualisierung von dreidimensionalen Volumensdaten aus dem medizinischen Bereich (YaDiV ) integriert.
Beschreibungstext verbergen
Betreuer: Karl-Ingo Friese April 2009
Oktober 2009
|
||||||
|
Seminararbeit
Marc Christoph Vollmer
Leibniz Universität Hannover
Kurzvortrag: Entwurf und Implementation einer haptischen Schnittstelle
Beschreibungstext anzeigen
Marc Christoph Vollmer hält einen Kurzvortrag zum Beginn seiner Masterarbeit mit dem Titel "Entwurf und Implementation einer haptischen Schnittstelle für YaDiV"
Im Rahmen dieser Arbeit werden Methoden zur effektiven und stabilen Integration eines haptischen Eingabegeräts in das Programm YaDiv untersucht. Hinsichtlich der bisher zur Verfügung stehenden Eingabegeräte werden Mechanismen zur taktilen Wahrnehmung nicht behandelt. Diese Arbeit beschränkt ausschließlich auf die kinästhetische Wahrnehmung.
Beschreibungstext verbergen
Mit der hier entwickelten haptischen Schnittstelle soll dem Benutzer die Möglichkeit gegeben werden, unter Verwendung eines entsprechenden Eingabegeräts, effektiver und realistischer im dreidimensionalen VR-Raum zu navigieren als es bisher mit einer zweidimensionalen Maus möglich ist. Neben dem haptischen Eingabegeräten wird auch die Erstellung eines Prototypen zur Benutzung eines Datenhandschuhs Bestandteil sein. Betreuer: Karl-Ingo Friese Mai 2009
|
||||||
|
Masterarbeit
Maximilian Müller
Leibniz Universität Hannover
High-Quality Ray Casting Verfahren zur Visualisierung segmentierter medizinischer Voxeldaten
Beschreibungstext anzeigen
Betreuer: Karl-Ingo Friese Oktober 2008
April 2009
|
||||||
| 2008 | ||||||
|
Studienarbeit
Lara Toma
Untersuchung zur Grauwertdarstellung auf LC-Displays
Beschreibungstext anzeigen
In der medizinische Diagnostik werden bei der Röntgenbildaufnahme oder Tomographie hingegen spezielle Graustufenmonitore ("Befundungsmonitore") verwendet, die bis zu 4096 unterschiedliche Graustufen darstellen können um z.B. Röntgenbilder oder CT-Aufnahmen beurteilen zu können. Dabei gilt schwarz als niedrigster und weiß als höchster Absoptionswert. Ein handelsüblicher PC kann auf einem RGB Monitor insgesamt 16,8 Millionen Farben darstellen, aber nur 256 Graustufen. Hier stellt sich die Frage, ob es wirklich nötig ist, mehr als 256 Abstufungen am Monitor darzustellen. Werden die Graustufen von normalen 8-Bit Monitoren genauso gut dargestellt, oder ist die Abstufung von 256 Grautönen für medizinische Anwendungen ein Muss, weil sonst 8-bit-Displays zu Fehldiagnosen oder unentdeckten Details führen könnten? Um diese Fragen beantworten zu können, wurden in dieser Studienarbeit unterschiedliche Versuche durchgeführt. Es wurde untersucht, wieviele Betreuer: Karl-Ingo Friese März 2008
Juni 2008
|
||||||
| 2007 | ||||||
|
Diplomarbeit
Dominik Sarnow
Leibniz Universität Hannover
Analyse von Direct Volume Rendering Verfahren zur Visualisierung von Voxeldaten
Beschreibungstext anzeigen
Inhalt dieser Arbeit war die Implementierung und der Vergleich verschiedener Direct Volume Rendering Verfahren zur Visualisierung von Voxeldaten. In der Medizin werden 3D-Bildgebende Verfahren wie die Computertomographie (CT) täglich eingesetzt, um eine digitale Repräsentation des Menschlichen Körpers oder Körperteilen zu gewinnen. Diese Daten werden üblicherweise als Voxeldaten gespeichert. Einen Voxel kann man sich als kleinen "Würfel" (quasi ein dreidimensionales Gegenstück zum 2D-Pixel) vorstellen. Jeder Voxel enthält einen zusätzlichen Wert, der bei der Computertomographie als Materialdichte interpretiertwerden kann - und bei der Visualisierung üblicherweise als Graustufe dargestellt wird. In dieser Arbeit werden verschiedene Verfahren Voxeldaten zu visualisieren verglichen. Diese Verfahren lassen sich grob in 4 Kategorien einteilen.
Ziel der Arbeit war es, mindestens ein Verfahren aus aus jeder Kategorie zu implementieren und hinsichtlich Laufzeit, Speicherverbrauch und optischem Eindruck zu vergleichen. Übergeordnetes Projekt: YaDiV Betreuer: Karl-Ingo Friese Juni 2007
Dezember 2007
|
||||||
|
Masterarbeit
Richard Guercke
Leibniz Universität Hannover
Boundary Extraction from Voxel Data Using Contour Lines
Beschreibungstext anzeigen
 In this thesis, an algorithm is introduced that extracts a boundary surface from a voxel data set representation of a shape. To achieve this objective, a connectivity graph is constructed from boundary polygons of the object in (usually orthogonal) 2D slices. This graph is used to find facets of the object's boundary surface. In this thesis, an algorithm is introduced that extracts a boundary surface from a voxel data set representation of a shape. To achieve this objective, a connectivity graph is constructed from boundary polygons of the object in (usually orthogonal) 2D slices. This graph is used to find facets of the object's boundary surface.At given sampling intervals, the resulting triangulated surfaces bear a considerably closer resemblance to the original voxel data object than the result of the (modified) Marching Cubes algorithm. This is, however, only achieved at the cost of an increase in computation time and memory usage. At the closest sampling interval, the algorithm returns a closed surface without self-intersections. The connectivity graph can also be used for the restriction of surface patches to be displayed according to visibility or as a basis for physical computations like FEM analysis. Project: YaDiV Betreuer: Karl-Ingo Friese Juni 2007
November 2007
|
||||||
|
Bachelorarbeit
Marcus Rieche
Leibniz Universität Hannover
OpenSG - Verteilte Visualisierung in verschiedenen Anwendungsszenarien
Beschreibungstext anzeigen
Die 3D Grafikhardware hat gerade bei Stanard PCs in den letzten Jahren große Fortschritte gemacht. Dennoch gibt es auch heute noch Grenzen, z.B. bei der Visualisierung großer Voxel-Daten aus dem medizinischen Bereich. Abhilfe schaffen entweder teure Spezialcomputer (Graphic Workstations) oder ein verteilter Ansatz, auch Clustering genannt.
Beschreibungstext verbergen
Betreuer: Karl-Ingo Friese Februar 2007
Juni 2007
|
||||||
|
Diplomarbeit
Yifan Yu
Leibniz Universität Hannover
Algorithmen zur Randflächenapproximation von Volumenkörpern in Voxeldarstellung
Beschreibungstext anzeigen
 Volumenvisualisierung beschreibt Verfahren, dreidimensionale Volumenkörper, beispielsweise durch Voxeldaten beschrieben, in geeigneter Art und Weise auf einem Computerbildschirm sichtbar zu machen. Dadurch wird es möglich, abstrakten Datenmengen als dreidimensionalen Objekte, die die Daten beschreiben, in einer virtuellen Welt realistisch darzustellen. Dabei kann man anschaulich mit den Daten arbeiten: z.B. die Oberfläche bzw. eine Isofläche berechnen, nach benutzerdefinierten Werten segmentieren, lokale Teilgebiete genauer untersuchen usw. Das Thema findet in vielen wissenschaftlichen und technischen Bereichen Anwendung: Geowissenschaft, Astrophysik, Chemie, Mikroskopie, Maschinenbau, zerstörungsfreie Prüfung usw. In dieser Diplomarbeit wird die Anwendung im Bereich der Medizin betrachtet.
In dieser Diplomarbeit wird die Anwendung im Bereich der Medizin betrachtet. Medizinische Daten über Patienten bestehen aus Materialdichte, die z.B. durch CT (Computertomographie), MRT (Magnetresonanztomographie) o.ä. gewonnen werden, wobei das zu untersuchende Objekt von den Geräten in regelmäßigen Abständen gemessen wird. Volumenvisualisierungstechniken werden in der Regel in zwei Kategorien eingeteilt: direktes Volumenrendering sowie Rekonstruktion der Oberfläche. Thema dieser Diplomarbeit ist die Oberflächenrekonstruktion.
Es gibt zahlreiche Methoden für die Rekonstruktion einer Oberfläche. Sie haben jeweils Vorteile und Nachteile und eignen sich daher für verschiedene Aufgaben. In meiner Diplomarbeit habe ich drei verschiedenen Gruppen untersucht: Marching Cubes, Contour Lines und GDM (Geometrically Deformed Models). In dieser Arbeit wurden Verfahren aus den ersten beiden Kategorien implementiert und auf medizinische Daten angewendet, um zu analysieren und zu vergleichen, wie gut sie in verschiedener Hinsicht geeignet sind. Volumenvisualisierung beschreibt Verfahren, dreidimensionale Volumenkörper, beispielsweise durch Voxeldaten beschrieben, in geeigneter Art und Weise auf einem Computerbildschirm sichtbar zu machen. Dadurch wird es möglich, abstrakten Datenmengen als dreidimensionalen Objekte, die die Daten beschreiben, in einer virtuellen Welt realistisch darzustellen. Dabei kann man anschaulich mit den Daten arbeiten: z.B. die Oberfläche bzw. eine Isofläche berechnen, nach benutzerdefinierten Werten segmentieren, lokale Teilgebiete genauer untersuchen usw. Das Thema findet in vielen wissenschaftlichen und technischen Bereichen Anwendung: Geowissenschaft, Astrophysik, Chemie, Mikroskopie, Maschinenbau, zerstörungsfreie Prüfung usw. In dieser Diplomarbeit wird die Anwendung im Bereich der Medizin betrachtet.
In dieser Diplomarbeit wird die Anwendung im Bereich der Medizin betrachtet. Medizinische Daten über Patienten bestehen aus Materialdichte, die z.B. durch CT (Computertomographie), MRT (Magnetresonanztomographie) o.ä. gewonnen werden, wobei das zu untersuchende Objekt von den Geräten in regelmäßigen Abständen gemessen wird. Volumenvisualisierungstechniken werden in der Regel in zwei Kategorien eingeteilt: direktes Volumenrendering sowie Rekonstruktion der Oberfläche. Thema dieser Diplomarbeit ist die Oberflächenrekonstruktion.
Es gibt zahlreiche Methoden für die Rekonstruktion einer Oberfläche. Sie haben jeweils Vorteile und Nachteile und eignen sich daher für verschiedene Aufgaben. In meiner Diplomarbeit habe ich drei verschiedenen Gruppen untersucht: Marching Cubes, Contour Lines und GDM (Geometrically Deformed Models). In dieser Arbeit wurden Verfahren aus den ersten beiden Kategorien implementiert und auf medizinische Daten angewendet, um zu analysieren und zu vergleichen, wie gut sie in verschiedener Hinsicht geeignet sind. Betreuer: Karl-Ingo Friese Februar 2007
|
||||||
| 2006 | ||||||
|
Masterarbeit
Marc Herrlich
Leibniz Universität Hannover
Untersuchungen zur Eignung von 3D-Spieleengines für die wissenschaftliche Visualisierung anhand der CryENGINE
Beschreibungstext anzeigen
 Computerspiele sind in heutiger Zeit aus dem Alltag vieler Menschen nicht mehr wegzudenken. Darüber hinaus erreichte die Technologie moderner Spiele im Laufe der letzten Jahre einen Standard, der diese auch für Anwendungen fernab des Unterhaltungssektors interessant macht.
Die Visualisierung wissenschaftlicher Daten ist ein bedeutendes Anwendungsgebiet der graphischen Datenverarbeitung. Insbesondere die interaktive dreidimensionale Darstellung virtueller oder realer Umgebungen stellt ein wichtiges Hilfsmittel dar.
In dieser Arbeit wird anhand der in dem Computerspiel Farcry eingesetzten CryEngine untersucht, welche Möglichkeiten moderne Spieleengines für die wissenschaftliche Arbeit bieten. Als Anwendungsbeispiel soll mit Hilfe der CryEngine eine auf GIS-Daten basierende dreidimensionale Landschaftsdarstellung umgesetzt werden. Dazu wird in der vorliegenden Arbeit ein Konverter entwickelt, um automatisiert aus vorhandenen GIS-Daten einen entsprechenden Farcry-Level zu erzeugen.
Computerspiele sind in heutiger Zeit aus dem Alltag vieler Menschen nicht mehr wegzudenken. Darüber hinaus erreichte die Technologie moderner Spiele im Laufe der letzten Jahre einen Standard, der diese auch für Anwendungen fernab des Unterhaltungssektors interessant macht.
Die Visualisierung wissenschaftlicher Daten ist ein bedeutendes Anwendungsgebiet der graphischen Datenverarbeitung. Insbesondere die interaktive dreidimensionale Darstellung virtueller oder realer Umgebungen stellt ein wichtiges Hilfsmittel dar.
In dieser Arbeit wird anhand der in dem Computerspiel Farcry eingesetzten CryEngine untersucht, welche Möglichkeiten moderne Spieleengines für die wissenschaftliche Arbeit bieten. Als Anwendungsbeispiel soll mit Hilfe der CryEngine eine auf GIS-Daten basierende dreidimensionale Landschaftsdarstellung umgesetzt werden. Dazu wird in der vorliegenden Arbeit ein Konverter entwickelt, um automatisiert aus vorhandenen GIS-Daten einen entsprechenden Farcry-Level zu erzeugen.Betreuer: Karl-Ingo Friese März 2006
|
||||||
|
Studienarbeit
Stefan Huy
Leibniz Universität Hannover
Design und Implementierung eines Objektmodells zur Erstellung und Verwendung komplexer Grafikobjekte in Webseiten
Beschreibungstext anzeigen
Web pages traditionally only allow the on-line manipulation of text items. To enable the web user to use graphics in web pages as a medium for discussion and presentation of concepts an object model is presented that allows the declarative description and in-page construction of composite pictures that can be used like normal images by integrating the delivered interpreter engine. The current implementation relies on a prototype based approach and uses a HTML and Javascript based file format as the descriptor. The open approach furthermore allows the easy implementation of the algorithm in other programming languages and therefore the integration into existing systems.
Beschreibungstext verbergen
Betreuer: Karl-Ingo Friese Januar 2006
|
||||||
| 2005 | ||||||
|
Studienarbeit
Dominik Sarnow
Leibniz Universität Hannover
3D Welten - Eine Untersuchung zur Visualisierung wissenschaftlicher Daten mit Spiele-Engines
Beschreibungstext anzeigen
Die Unterhaltungsindustrie ist ein grosser und wichtiger Markt geworden und es fließen beachtliche Summen in die Entwicklung neuer Game Engines. Ausserdem, und das ist ein entscheidender Vorteil der Game Engines gegenüber anderen 3D-Visualisierungsprogrammen, ist die Anschaffung der Software wesentlich günstiger. Der Gedanke liegt also nahe, dieses Potential zu nutzen.
Beschreibungstext verbergen
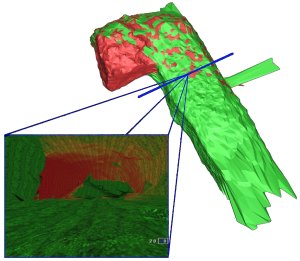 Am Institut für Mensch-Maschine-Kommunikation, Fachbereich Graphische Datenverarbeitung wurden in Zusammenarbeit mit dem Institut für Bergbau und Vermessungstechnik einsturzgefährdete Höhlen mit Hilfe eines Lasers vermessen.
In zwei darauf folgenden Diplomarbeiten von Bijan Frajollahi und Karl-Ingo Friese, wurden aus den Punktwolken, die eigentlichen Höhlen in Form von Dreiecksflächen, so genannten Triangle Mesh rekonstruiert. Es gab mehrere Messungen an verschiedenen Punkten in der Höhle und mit Hilfe eines Visualisierungsprogrammes konnten sie dargestellt werden.
Ziel und Motivation dieser Studienarbeit ist es nun an die voran gegangenen Schritte anzuknüpfen und die Höhlen begehbar zu machen, mit Hilfe einer der heutigen kommerziellen Game Engines. Bei der Rekonstruktion war das Augenmerk auf eine möglichst genaue, detaillierte Darstellung der Höhlen gerichtet. Hierfür war es unter anderem notwendig die Datenmenge zu verkleinern, (d.h. die Anzahl der Dreiecke reduzieren) ohne die Topologie zu verändern.
Links:
Am Institut für Mensch-Maschine-Kommunikation, Fachbereich Graphische Datenverarbeitung wurden in Zusammenarbeit mit dem Institut für Bergbau und Vermessungstechnik einsturzgefährdete Höhlen mit Hilfe eines Lasers vermessen.
In zwei darauf folgenden Diplomarbeiten von Bijan Frajollahi und Karl-Ingo Friese, wurden aus den Punktwolken, die eigentlichen Höhlen in Form von Dreiecksflächen, so genannten Triangle Mesh rekonstruiert. Es gab mehrere Messungen an verschiedenen Punkten in der Höhle und mit Hilfe eines Visualisierungsprogrammes konnten sie dargestellt werden.
Ziel und Motivation dieser Studienarbeit ist es nun an die voran gegangenen Schritte anzuknüpfen und die Höhlen begehbar zu machen, mit Hilfe einer der heutigen kommerziellen Game Engines. Bei der Rekonstruktion war das Augenmerk auf eine möglichst genaue, detaillierte Darstellung der Höhlen gerichtet. Hierfür war es unter anderem notwendig die Datenmenge zu verkleinern, (d.h. die Anzahl der Dreiecke reduzieren) ohne die Topologie zu verändern.
Links:
Betreuer: Karl-Ingo Friese Juni 2005
|
||||||
|
Bachelorarbeit
Daniela Lauer
Leibniz Universität Hannover
Zerlegung polygonal berandeter Gebiete in der euklidischen Ebene in konvexe Teilbereiche
Beschreibungstext anzeigen
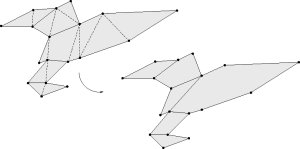 In der Computergrafik werden möglichst einfache Darstellungen verwendet um geometrische Objekte zu modellieren. Eine gängige Methode ist es ein Objekt durch die Approximation seiner Oberfläche darzustellen. Dabei wird diese aus einer Menge planarer Vielecke,
hier Polygone genannt, zusammengesetzt. Damit auch komplexe Objekte effektiv verwaltet und bearbeitet werden können, wird jedoch noch eine einfachere Darstellung der Teilstücke benötigt. Eine Möglichkeit ist, Polygone als die Vereinigung von weniger komplexen Teilstücken zu repräsentieren. Ein konvex zerlegtes Polygon setzt sich nur aus konvexen Teilstücken (einfacheren Polygonen) zusammen.
Die Motivation für die vorliegende Arbeit kommt aus der Computergrafik aus dem Gebiet wissenschaftliche Visualisierung mit Spiele-Engines. Für eine optisch anspruchsvolle Visualisierung in Echtzeit wird unter anderem eine leistungsstarke Renderingsoftware benötigt. Eine günstige Alternative zu kommerziellen Produkten wie z.B. 3D Studio MAX oder Autocad bieten Spiele-Engines. Im Gegensatz zu den Anfängen der Computergrafik sind heute die Hersteller von animierten Filmen und Computerspielen führend im Bereich der Echtzeit 3D-Visualisierung.
Deshalb wird am Lehrstuhl Graphische Datenverarbeitung, Institut für Mensch-Maschine-Kommunikation der Leibniz Universität Hannover, die Eignung von Spieleengines für die Visualisierung wissenschaftlicher Daten anhand einiger Beispiele (Sarnow: Quake 3, Hanel: Unreal 2) erprobt. Die von einem Laserscanner gemessene Punktwolke der Höhlenwände wurde in Diplomarbeiten (Farajollahi, Friese,) in eine triangulierte Darstellung überführt und dient als Datenquelle für diese Untersuchungen. Erste Ergebnisse ermöglichen ein flüssiges Durchwandern der Höhle. In der Darstellung mit der Quake-Engine führte jedoch beispielsweise das Hinzufügen von aufwändigen Lichteffekten zu Performanceverlusten. Dies liegt unter anderem an der Menge der darzustellende Polygone (bislang Dreiecke). Die Quake Engine ist jedoch in der Lage, nicht nur Dreiecke, sondern konvexe Polygone einzulesen und zu verarbeiten. Dies eröffnet die Möglichkeit, durch das Verwenden größerer konvexer Teilstücke mit möglichst wenig Detailverlust die Anzahl der Polygone zu reduzieren.
Die Idee liegt also nahe, nach großen planaren oder quasiplanaren Teilgebieten bestehend aus mehreren Dreiecken zu suchen, um diese dann als Vereinigung einer Menge von konvexen Polygonen darzustellen. Es wird also ein Algorithmus benötigt, der ein in triangulierter Form vorliegendes Gebiet (Polygone) in möglichst wenige konvexe Teilstücke zerlegt.
In der Computergrafik werden möglichst einfache Darstellungen verwendet um geometrische Objekte zu modellieren. Eine gängige Methode ist es ein Objekt durch die Approximation seiner Oberfläche darzustellen. Dabei wird diese aus einer Menge planarer Vielecke,
hier Polygone genannt, zusammengesetzt. Damit auch komplexe Objekte effektiv verwaltet und bearbeitet werden können, wird jedoch noch eine einfachere Darstellung der Teilstücke benötigt. Eine Möglichkeit ist, Polygone als die Vereinigung von weniger komplexen Teilstücken zu repräsentieren. Ein konvex zerlegtes Polygon setzt sich nur aus konvexen Teilstücken (einfacheren Polygonen) zusammen.
Die Motivation für die vorliegende Arbeit kommt aus der Computergrafik aus dem Gebiet wissenschaftliche Visualisierung mit Spiele-Engines. Für eine optisch anspruchsvolle Visualisierung in Echtzeit wird unter anderem eine leistungsstarke Renderingsoftware benötigt. Eine günstige Alternative zu kommerziellen Produkten wie z.B. 3D Studio MAX oder Autocad bieten Spiele-Engines. Im Gegensatz zu den Anfängen der Computergrafik sind heute die Hersteller von animierten Filmen und Computerspielen führend im Bereich der Echtzeit 3D-Visualisierung.
Deshalb wird am Lehrstuhl Graphische Datenverarbeitung, Institut für Mensch-Maschine-Kommunikation der Leibniz Universität Hannover, die Eignung von Spieleengines für die Visualisierung wissenschaftlicher Daten anhand einiger Beispiele (Sarnow: Quake 3, Hanel: Unreal 2) erprobt. Die von einem Laserscanner gemessene Punktwolke der Höhlenwände wurde in Diplomarbeiten (Farajollahi, Friese,) in eine triangulierte Darstellung überführt und dient als Datenquelle für diese Untersuchungen. Erste Ergebnisse ermöglichen ein flüssiges Durchwandern der Höhle. In der Darstellung mit der Quake-Engine führte jedoch beispielsweise das Hinzufügen von aufwändigen Lichteffekten zu Performanceverlusten. Dies liegt unter anderem an der Menge der darzustellende Polygone (bislang Dreiecke). Die Quake Engine ist jedoch in der Lage, nicht nur Dreiecke, sondern konvexe Polygone einzulesen und zu verarbeiten. Dies eröffnet die Möglichkeit, durch das Verwenden größerer konvexer Teilstücke mit möglichst wenig Detailverlust die Anzahl der Polygone zu reduzieren.
Die Idee liegt also nahe, nach großen planaren oder quasiplanaren Teilgebieten bestehend aus mehreren Dreiecken zu suchen, um diese dann als Vereinigung einer Menge von konvexen Polygonen darzustellen. Es wird also ein Algorithmus benötigt, der ein in triangulierter Form vorliegendes Gebiet (Polygone) in möglichst wenige konvexe Teilstücke zerlegt.Betreuer: Karl-Ingo Friese Januar 2005
|
||||||
| 2004 | ||||||
|
Bachelorarbeit
Michael Hanel
Leibniz Universität Hannover
3D-Visualisierung mit Hilfe von Spiel-Engines unter Berücksichtigung erweiterter Texturierungsmöglichkeiten
Beschreibungstext anzeigen
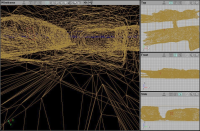 Nowadays, the graphical (re)presentation of several computer-based applications becomes more important, especially in the entertainment electronics sector.
With the Unreal Engine 2 and the associated application Unreal Tournament 2004, the user has the possibility to change some parameters of the engine (so called Modifications) and to create his own maps.
Nowadays, the graphical (re)presentation of several computer-based applications becomes more important, especially in the entertainment electronics sector.
With the Unreal Engine 2 and the associated application Unreal Tournament 2004, the user has the possibility to change some parameters of the engine (so called Modifications) and to create his own maps.
If we try to use the Unreal Engine 2 to visualize scientific data (e.g. a scatter-plot of a cave obtained from a laser measurement), the problem of how the data has to be formatted, so that the engine is able to read it, arises. In this Bachelor-thesis, the approach of representing scientific data with the Unreal Engine 2 as realistic as possible is being accomplished. Betreuer: Karl-Ingo Friese Dezember 2004
|



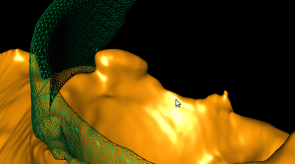
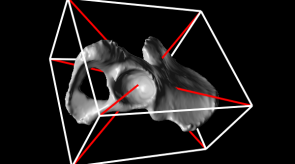
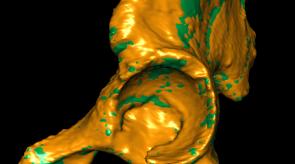
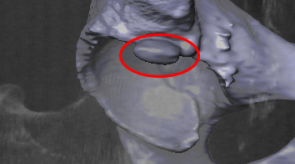
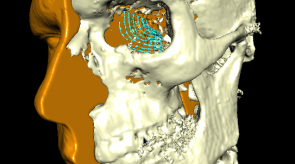
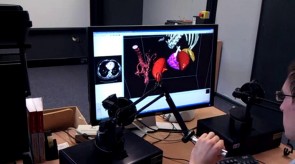

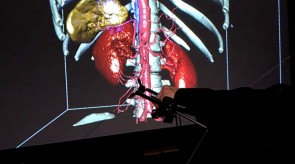
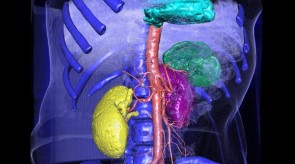
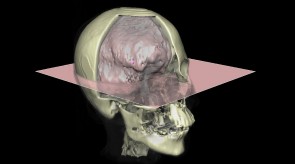
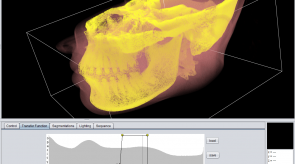




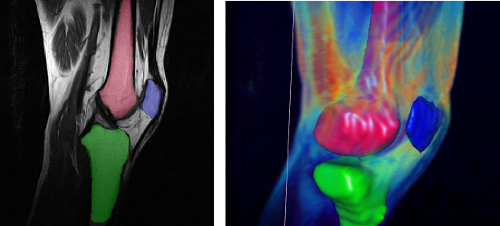
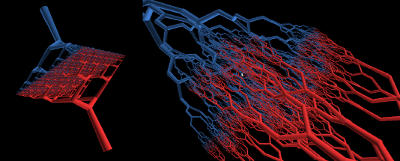

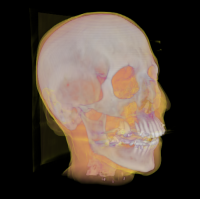 Bildgebende Verfahren haben insbesondere in der medizinischen Anwendung eine große Verbreitung erfahren. So sind z.B. Computertomographie oder Magnet-Resonanz-Tomographie aus dem klinsichen Alltag nicht mehr wegzudenken. Um die gewonnen 3D-Daten auch 3-dimensional zu visualiseren, können unterschiedliche Methoden des Direct-Volume-Rendering verwendet werden. Die Algorithmen unterscheiden sich vor allem in Laufzeit und Bildqualität. Ein vergleichender Überblick über die gängigen Verfahren wird z.B. in der Diplomarbeit von Dominik Sarnow gegeben.
Bildgebende Verfahren haben insbesondere in der medizinischen Anwendung eine große Verbreitung erfahren. So sind z.B. Computertomographie oder Magnet-Resonanz-Tomographie aus dem klinsichen Alltag nicht mehr wegzudenken. Um die gewonnen 3D-Daten auch 3-dimensional zu visualiseren, können unterschiedliche Methoden des Direct-Volume-Rendering verwendet werden. Die Algorithmen unterscheiden sich vor allem in Laufzeit und Bildqualität. Ein vergleichender Überblick über die gängigen Verfahren wird z.B. in der Diplomarbeit von Dominik Sarnow gegeben.
